Estructura poblacional y diversidad genética de una pequeña población de ganado lechero de raza Montbéliarde en Ecuador
Contenido principal del artículo
Resumen
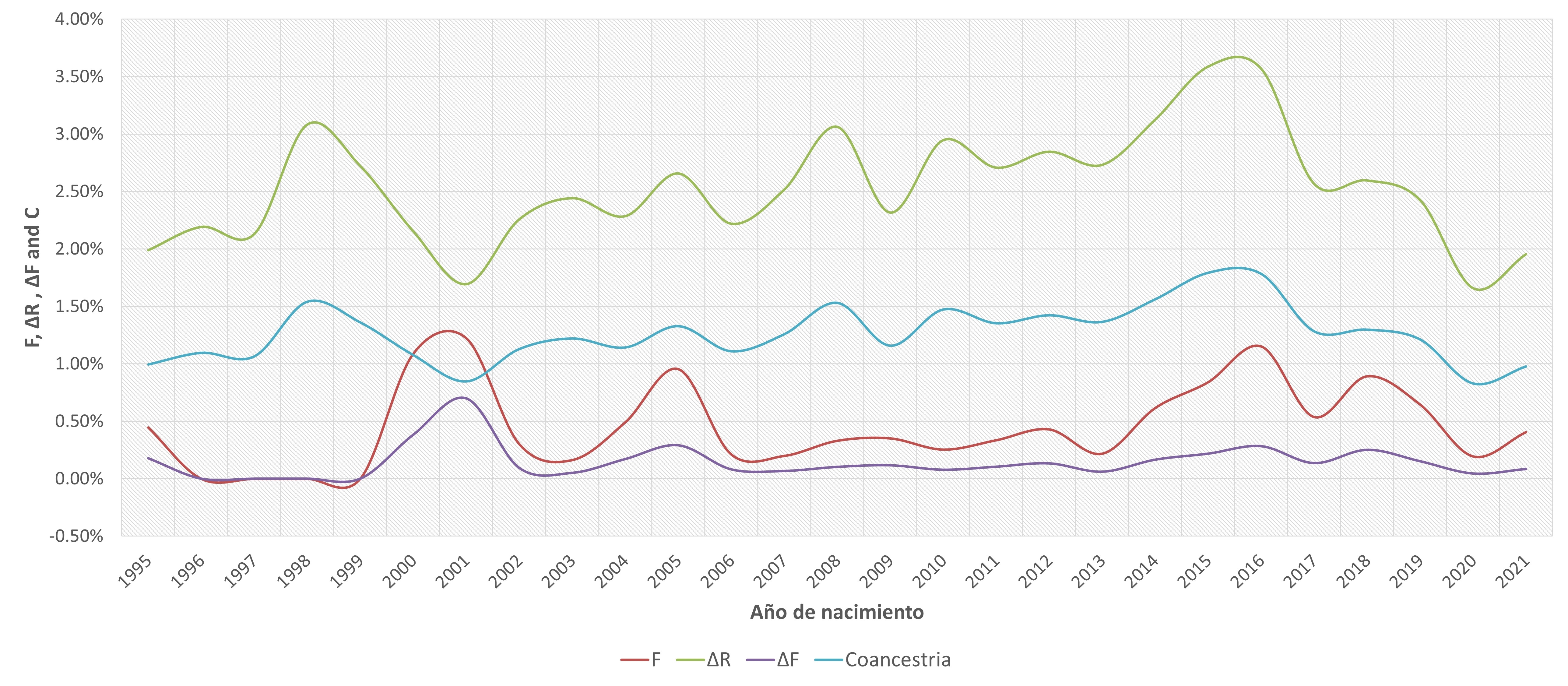
En Ecuador, la Asociación Montbéliarde ha utilizado el método de cruzamiento por absorción, para la obtención de crías puras debido a las dificultades y costos elevados de otras opciones. El objetivo de este estudio fue analizar la diversidad genética y la estructura de la población registrada, para esto se consideraron los datos de la Asociación Montbéliarde constituida por 210 toros y 1584 vacas. La base datos fue dividida en tres grupos uno histórico, uno actual y uno de referencia. Se estimaron parámetros de estructura y diversidad genética complementados con un análisis de probabilidad de origen de genes. Para los cálculos se utilizó el programa ENDOG. La estructura y diversidad genética de la población Montbéliarde presentó valores adecuados para el desarrollo de un plan de mejora genética y tendría un futuro prometedor si se implementara políticas más integrales en la cría.
Descargas
Métricas
Detalles del artículo

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Los autores que publican en Siembra conocen y aceptan las siguientes condiciones:
- Los autores retienen los derechos de copia (copyright) y ceden a Siembra el derecho de primera publicación del trabajo, bajo licencia Creative Commons Attribution License, que permite a terceros utilizar lo publicado siempre que hagan referencia al autor o autores del trabajo y a su publicación en esta revista.
![]() Esta obra está bajo una Licencia Creative Commons Reconocimiento-NoComercial 4.0 Internacional (CC BY-NC 4.0).
Esta obra está bajo una Licencia Creative Commons Reconocimiento-NoComercial 4.0 Internacional (CC BY-NC 4.0).
- Los autores conservan los derechos de autor y garantizan a Siembra el derecho de publicar el manuscrito a través de los canales que considere adecuados.
- Los autores pueden establecer por su cuenta acuerdos adicionales para la distribución no exclusiva de la versión de la obra publicada en Siembra, haciendo reconocimiento de su publicación inicial en la misma, como por ejemplo en repositorios institucionales.
Se autoriza a los autores a difundir sus trabajos electrónicamente una vez sea aceptado el manuscrito para su publicación.
Citas
Ablondi, M., Summer, A., Stocco, G., Finocchiaro, R., van Kaam, J.-T., Cassandro, M., Dadousis, C., Sabbioni, A., y Cipolat-Gotet, C. (2023). The role of inbreeding depression on productive performance in the Italian Holstein breed. Journal of Animal Science, 101, skad382. https://doi.org/10.1093/jas/skad382 DOI: https://doi.org/10.1093/jas/skad382
Barbat, A., Mézec, P. Le, Ducrocq, V., Mattalia, S., Fritz, S., Boichard, D., Ponsart, C., y Humblot, P. (2010). Female fertility in French dairy breeds: current situation and strategies for improvement. Journal of Reproduction and Development, 56(S), S15-S21. https://doi.org/10.1262/jrd.1056S15 DOI: https://doi.org/10.1262/jrd.1056S15
Boichard, D., Guillaume, F., Baur, A., Croiseau, P., Rossignol, M. N., Boscher, M. Y., Druet, T., Genestout, L., Colleau, J. J., Journaux, L., Ducrocq, V., y Fritz, S. (2012). Genomic selection in French dairy cattle. Animal Production Science, 52(3), 115. https://doi.org/10.1071/AN11119 DOI: https://doi.org/10.1071/AN11119
Boichard, D., Maignel, L., y Verrier, É. (1997). The value of using probabilities of gene origin to measure genetic variability in a population. Genetics Selection Evolution, 29(1), 5. https://doi.org/10.1186/1297-9686-29-1-5 DOI: https://doi.org/10.1051/gse:19970101
Bonilla Jurado, A. G. (2020). Evaluación de las transferencias realizadas por el gobierno central al sector lechero ecuatoriano durante el período 2012-2017. En Nuevos aportes a la economía. Especialidad: Economía Social de Mercado (pp. 9-122). Editorial PUCE. https://latinamerica.hss.de/fileadmin/user_upload/Projects_HSS/Latin_America/Ecuador/Dokumente/2020/Libro_PUCE_2020.pdf
Caballero, A., y Toro, M. A. (2000). Interrelations between effective population size and other pedigree tools for the management of conserved populations. Genetical Research, 75(3), 331-343. https://doi.org/10.1017/S0016672399004449 DOI: https://doi.org/10.1017/S0016672399004449
Cartuche Macas, L. F., Camacho Vallejo, M. E., González Ariza, A., León Jurado, J. M., Delgado Bermejo, J. V., Marín Navas, C., y Navas González, F. J. (2024a). Analysis of endangered Andalusian Black cattle (Negra Andaluza) reveals genetic reservoir for bovine black trunk. Animals (Basel), 14(7), 1131. https://doi.org/10.3390/ani14071131 DOI: https://doi.org/10.3390/ani14071131
Cartuche, L., Vargas, N., y Pascual, M. (2014). Análisis preliminar del pedigrí de las razas bovinas lecheras Jersey y Brown Swiss en el Ecuador. En IX Congreso de Ciencia y Tecnología ESPE 2014. Quito. https://journal.espe.edu.ec/ojs/index.php/cienciaytecnologia/article/view/79/77
Cartuche-Macas, L. F., Lozada, E. F., Gutiérrez-Reinoso, M. A., Chacón, E., Navas, F. J., y García-Herreros, M. (2024b). Evolution of population structure, reproductive performance, inbreeding, and genetic diversity in Ecuadorian Charolais Cattle. Veterinary Sciences, 11(11), 566. https://doi.org/10.3390/vetsci11110566 DOI: https://doi.org/10.3390/vetsci11110566
Casanovas Arias, D., León Jurado, J. M., Bermejo Asensio, L. A., Navas González, F. J., Marín Navas, C., y Barba Capote, C. J. (2020). Genetic diversity evolution of a sheep breed reintroduced after extinction: Tracing back Christopher Columbus’ first imported sheep. Research in Veterinary Science, 132, 207-216. https://doi.org/10.1016/j.rvsc.2020.06.007 DOI: https://doi.org/10.1016/j.rvsc.2020.06.007
Cervantes, I., Goyache, F., Molina, A., Valera, M., y Gutiérrez, J. P. (2011). Estimation of effective population size from the rate of coancestry in pedigreed populations. Journal of Animal Breeding and Genetics, 128(1), 56–63. https://doi.org/10.1111/j.1439-0388.2010.00881.x DOI: https://doi.org/10.1111/j.1439-0388.2010.00881.x
Danchin‐Burge, C., Leroy, G., Brochard, M., Moureaux, S., y Verrier, E. (2012). Evolution of the genetic variability of eight French dairy cattle breeds assessed by pedigree analysis. Journal of Animal Breeding and Genetics, 129(3), 206-217. https://doi.org/10.1111/j.1439-0388.2011.00967.x DOI: https://doi.org/10.1111/j.1439-0388.2011.00967.x
ESPAE Graduate School of Management. (2016). Estudios industriales orientación estratégica para la toma de decisiones: Industria de Ganadería de Carne. ESPOL. https://www.espae.edu.ec/publicaciones/estudios-industriales-orientacion-estrategica-para-la-toma-de-decisiones-industria-de-ganaderia-de-carne/
Falconer, D. S., y Mackay, T. F. C. (1996). Introduction to quantitative genetics (4th ed.). Longman.
García-Ruiz, A., Cole, J. B., VanRaden, P. M., Wiggans, G. R., Ruiz-López, F. J., y Van Tassell, C. P. (2016). Correction for Garcia-Ruiz et al., Changes in genetic selection differentials and generation intervals in US Holstein dairy cattle as a result of genomic selection. Proceedings of the National Academy of Sciences, 113(33), E4928. https://doi.org/10.1073/pnas.1611570113 DOI: https://doi.org/10.1073/pnas.1519061113
Gicquel, E., Boettcher, P., Besbes, B., Furre, S., Fernández, J., Danchin-Burge, C., Berger, B., Baumung, R., Feijóo, J. R. J., y Leroy, G. (2020). Impact of conservation measures on demography and genetic variability of livestock breeds. Animal, 14(4), 670-680. https://doi.org/10.1017/S1751731119002672 DOI: https://doi.org/10.1017/S1751731119002672
González, A. R. M., Navas González, F. J., Crudeli, G. Á., Delgado Bermejo, J. V., Camacho Vallejo, M. E., y Quirino, C. R. (2022). Process of introduction of Australian Braford cattle to South America: configuration of population structure and genetic diversity evolution. Animals, 12(3), 275. https://doi.org/10.3390/ani12030275 DOI: https://doi.org/10.3390/ani12030275
Groeneveld, E., Westhuizen, B. V. D., Maiwashe, A., Voordewind, F., y Ferraz, J. B. S. (2009). POPREP: a generic report for population management. Genetics and Molecular Research, 8(3), 1158-1178. https://doi.org/10.4238/vol8-3gmr648 DOI: https://doi.org/10.4238/vol8-3gmr648
Gutiérrez, J. P., Cervantes, I., y Goyache, F. (2009). Improving the estimation of realized effective population sizes in farm animals. Journal of Animal Breeding and Genetics, 126(4), 327–332. https://doi.org/10.1111/j.1439-0388.2009.00810.x DOI: https://doi.org/10.1111/j.1439-0388.2009.00810.x
Gutiérrez, J. P., Marmi, J., Goyache, F., y Jordana, J. (2005). Pedigree information reveals moderate to high levels of inbreeding and a weak population structure in the endangered Catalonian donkey breed. Journal of Animal Breeding and Genetics, 122(6), 378-386. https://doi.org/10.1111/j.1439-0388.2005.00546.x DOI: https://doi.org/10.1111/j.1439-0388.2005.00546.x
Gutiérrez, J. P., y Goyache, F. (2005). A note on ENDOG: a computer program for analysing pedigree information. Journal of Animal Breeding and Genetics, 122(3), 172-176. https://doi.org/10.1111/j.1439-0388.2005.00512.x DOI: https://doi.org/10.1111/j.1439-0388.2005.00512.x
Hagger, C. (2005). Estimates of genetic diversity in the brown cattle population of Switzerland obtained from pedigree information. Journal of Animal Breeding and Genetics, 122(6), 405–413. https://doi.org/10.1111/j.1439-0388.2005.00552.x DOI: https://doi.org/10.1111/j.1439-0388.2005.00552.x
Honda, T., Nomura, T., Yamaguchi, Y., y Mukai, F. (2004). Monitoring of genetic diversity in the Japanese Black cattle population by the use of pedigree information. Journal of Animal Breeding and Genetics, 121(4), 242-252. https://doi.org/10.1111/j.1439-0388.2004.00452.x DOI: https://doi.org/10.1111/j.1439-0388.2004.00452.x
Institut de l'Élevage [IDELE]. (2021). Bilan de variabilité génétique à partir des données de généalogies Races bovines laitières. IDELE. https://idele.fr/detail-article/indicateurs-de-variabilite-genetique-races-bovines-edition-2021
Institut de l'Élevage [IDELE].. (2023). Bilan de variabilité génétique à partir des données de généalogies Races bovines laitières. IDELE. https://idele.fr/detail-article/rapport-varume-2023-des-races-bovines-laitieres
James, J. W. (1977). A note on selection differential and generation length when generations overlap. Animal Science, 24(1), 109-112. https://doi.org/10.1017/S0003356100039271 DOI: https://doi.org/10.1017/S0003356100039271
Lacy, R. C. (1989). Analysis of founder representation in pedigrees: Founder equivalents and founder genome equivalents. Zoo Biology, 8(2), 111-123. https://doi.org/10.1002/zoo.1430080203 DOI: https://doi.org/10.1002/zoo.1430080203
Lindgren, D., Gea, L., y Jefferson, P. (1996). Loss of genetic diversity monitored by status number. Silvae Genetica, 45(1), 52-60.
Lozada, E., Chacón, E., Sambache, E., Revelo, M., Gutiérrez, M., Delgado, J. V., Cartuche, L. F., y Navas, F. J. (2023). Diversidad genética y estructura de la población de la raza Charolais en Ecuador a través del pedigrí. Archivos de Zootecnia, 72(277), 32-42. https://doi.org/10.21071/az.v72i277.5699 DOI: https://doi.org/10.21071/az.v72i277.5699
Lozada-Soto, E. A., Maltecca, C., Lu, D., Miller, S., Cole, J. B., y Tiezzi, F. (2021). Trends in genetic diversity and the effect of inbreeding in American Angus cattle under genomic selection. Genetics Selection Evolution, 53(1), 50. https://doi.org/10.1186/s12711-021-00644-z DOI: https://doi.org/10.1186/s12711-021-00644-z
Luštrek, B., Vandenplas, J., Gorjanc, G., y Potočnik, K. (2021). Genomic evaluation of Brown Swiss dairy cattle with limited national genotype data and integrated external information. Journal of Dairy Science, 104(5), 5738-5754. https://doi.org/10.3168/jds.2020-19493 DOI: https://doi.org/10.3168/jds.2020-19493
MacCluer, J. W., Boyce, A. J., Dyke, B., Weitkamp, L. R., Pfenning, D. W., y Parsons, C. J. (1983). Inbreeding and pedigree structure in Standardbred horses. Journal of Heredity, 74(6), 394-399. https://doi.org/10.1093/oxfordjournals.jhered.a109824 DOI: https://doi.org/10.1093/oxfordjournals.jhered.a109824
Macor, L. (2013). Evaluación de la variabilidad genética mediante el número efectivo en Braford Argentino Universitat Politècnica de València. https://riunet.upv.es/handle/10251/37144
Magalhães Araújo da Silva, M. H., Malhado, C. H. M., Costa, J. L., Cobuci, J. A., Costa, C. N., y Carneiro, P. L. S. (2016). Population genetic structure in the Holstein breed in Brazil. Tropical Animal Health and Production, 48(2), 331-336. https://doi.org/10.1007/s11250-015-0956-7 DOI: https://doi.org/10.1007/s11250-015-0956-7
Meuwissen, T., y Luo, Z. (1992). Computing inbreeding coefficients in large populations. Genetics Selection Evolution, 24(4), 305. https://doi.org/10.1186/1297-9686-24-4-305 DOI: https://doi.org/10.1051/gse:19920402
Mihai, R., Mărginean, G. E., Marin, M. P., Hassan, A. A. M., Marin, I., Fîntîneru, G., y Vidu, L. (2020). Impact of precision livestock farming on welfare and milk production in Montbeliarde dairy cows. Scientific Papers. Series D. Animal Science, 63(2), 308-313. https://animalsciencejournal.usamv.ro/index.php/scientific-papers/past-issues?id=811
Ministerio de Agricultura, Ganadería, Acuacultura y Pesca [MAGAP]. (2016). La política agropecuaria ecuatoriana: hacia el desarrollo territorial rural sostenible: 2015-2025. I Parte. MAGAP. https://www.fao.org/faolex/results/details/es/c/LEX-FAOC183434/
Navas, F. J., Jordana, J., León, J. M., Barba, C., y Delgado, J. V. (2017). A model to infer the demographic structure evolution of endangered donkey populations. Animal, 11(12), 2129-2138. https://doi.org/10.1017/S1751731117000969 DOI: https://doi.org/10.1017/S1751731117000969
Núñez-Domínguez, R., Martínez-Rocha, R. E., Hidalgo-Moreno, J. A., Ramírez-Valverde, R., y García-Muñiz, J. G. (2020). Evaluation of the Romosinuano cattle population structure in Mexico using pedigree analysis. Revista Colombiana de Ciencias Pecuarias, 33(1), 44-59. https://doi.org/10.17533/udea.rccp.v32n4a05 DOI: https://doi.org/10.17533/udea.rccp.v32n4a05
Oldenbroek, J. K. (ed.). (2017). Genomic management of animal genetic diversity. Wageningen Academic. https://doi.org/10.3920/978-90-8686-850-6 DOI: https://doi.org/10.3920/978-90-8686-850-6
Pais Chanfrau, J. M., Núñez Pérez, J., Lara Fiallos, M. v., Rivera Intriago, L. M., Trujillo Toledo, L. E., y Cuaran Guerrero, M. J. (2017). Valorización del suero de leche: Una visión desde la biotecnología. Bionatura, 2(4), 468–476. https://doi.org/10.21931/RB/2017.02.04.11 DOI: https://doi.org/10.21931/RB/2017.02.04.11
Peixoto, M. G. C. D., Carvalho, M. R. S., Egito, A. A., Steinberg, R. S., Bruneli, F. Â. T., Machado, M. A., Santos, F. C., Rosse, I. C., y Fonseca, P. A. S. (2021). Genetic Diversity and Population Genetic Structure of a Guzerá (Bos indicus) Meta-Population. Animals, 11(4), 1125. https://doi.org/10.3390/ani11041125 DOI: https://doi.org/10.3390/ani11041125
Ríos Utrera, Á., Vega Murillo, V. E., Montaño Bermúdez, M., Martínez Velázquez, G., y Román Ponce, S. I. (2018). Genetic diversity assessment of the Mexican Simmental population through pedigree analysis. Revista Brasileira de Zootecnia, 47, e20160088. https://doi.org/10.1590/rbz4720160088 DOI: https://doi.org/10.1590/rbz4720160088
Rodríguez-Ramilo, S. T., Fernández, J., Toro, M. A., Hernández, D., y Villanueva, B. (2015). Genome-Wide Estimates of Coancestry, Inbreeding and Effective Population Size in the Spanish Holstein Population. PLOS ONE, 10(4), e0124157. https://doi.org/10.1371/journal.pone.0124157 DOI: https://doi.org/10.1371/journal.pone.0124157
Ron Garrido, L., Birchmeier, A. N., Munilla, S., y Cantet, R. J. C. (2008). Estimation of effective population size using bivariate discrete distributions for modeling family size in beef cattle. Livestock Science, 117(1), 43–51. https://doi.org/10.1016/j.livsci.2007.11.008 DOI: https://doi.org/10.1016/j.livsci.2007.11.008
Rosendo Ponce, A., Palacios Jiménez, A. L., Rosales Martínez, F., Torres Hernández, G., Ramírez Valverde, R., y Becerril Pérez, C. M. (2018). Genetic variability of Tropical Milking Criollo cattle of Mexico estimated from genealogy information. Revista Colombiana de Ciencias Pecuarias, 31(3), 196-203. https://doi.org/10.17533/udea.rccp.v31n3a04 DOI: https://doi.org/10.17533/udea.rccp.v31n3a04
Sargolzaei, M., Iwaisaki, H., y Collleau, J. J. (2006). CFC: A tool for monitoring genetic diversity. En Proceedings of the 8th World Congress on Genetics Applied to Livestock Production. Belo Horizonte. https://www.cabidigitallibrary.org/doi/full/10.5555/20063170110
Scott, B. A., Haile-Mariam, M., Cocks, B. G., y Pryce, J. E. (2021). How genomic selection has increased rates of genetic gain and inbreeding in the Australian national herd, genomic information nucleus, and bulls. Journal of Dairy Science, 104(11), 11832-11849. https://doi.org/10.3168/jds.2021-20326 DOI: https://doi.org/10.3168/jds.2021-20326
Sieklicki, M. de F., Mulim, H. A., Pinto, L. F. B., Valloto, A. A., y Pedrosa, V. B. (2020). Population structure and inbreeding of Holstein cattle in southern Brazil. Revista Brasileira de Zootecnia, 49. e20190052. https://doi.org/10.37496/rbz4920190052 DOI: https://doi.org/10.37496/rbz4920190052
Superintendencia de Control del Poder de Mercado. (2015). Informe del sector lácteo en Ecuador. INFORME SCPM-IGT-INAC-002-2019. https://www.fao.org/faolex/results/details/es/c/LEX-FAOC183434/
Tenhunen, S., Thomasen, J. R., Sørensen, L. P., Berg, P., y Kargo, M. (2024). Genomic analysis of inbreeding and coancestry in Nordic Jersey and Holstein dairy cattle populations. Journal of Dairy Science, 107(8), 5897-5912. https://doi.org/10.3168/jds.2023-24553 DOI: https://doi.org/10.3168/jds.2023-24553
Uemoto, Y., Suzuki, K., Yasuda, J., Roh, S., y Satoh, M. (2021). Evaluation of inbreeding and genetic diversity in Japanese Shorthorn cattle by pedigree analysis. Animal Science Journal, 92(1). https://doi.org/10.1111/asj.13643 DOI: https://doi.org/10.1111/asj.13643
Vásquez-Loaiza, M., Cruz-Méndez, A., Castro-Vásquez, R., Cascante-Segura, S., Camacho-Sandoval, J., y Domínguez-Viveros, J. (2021). Pedigree analysis and population structure of brahman cattle from Costa Rica. Chilean Journal of Agricultural & Animal Sciences, 37(2), 177-183. https://doi.org/10.29393/CHJAAS37-20PAMJ60020 DOI: https://doi.org/10.29393/CHJAAS37-20PAMJ60020
Vassallo, J. M., Diaz, C., y Garcia-Medina, J. R. (1986). A note on the population structure of the Avileña breed of cattle in Spain. Livestock Production Science, 15(3), 285-288. https://doi.org/10.1016/0301-6226(86)90035-7 DOI: https://doi.org/10.1016/0301-6226(86)90035-7
Wang, Y. (2015). Genetic and geographic diversity of Gyr (Bos Indicus) cattle in Brazil. Universität für Bodenkultur Wien. https://permalink.obvsg.at/bok/AC12619943
Wright, S. (1922). Coefficients of Inbreeding and Relationship. The American Naturalist, 56(645), 330-338. https://doi.org/10.1086/279872 DOI: https://doi.org/10.1086/279872