Caracterización molecular de Escherichia coli productora de β-lactamasas de espectro extendido: Un estudio comparativo entre poblaciones porcinas y humanas
Main Article Content
Abstract
Introducción
La resistencia a los antimicrobianos [RAM] representa una amenaza creciente para la salud global, donde la resistencia a cefalosporinas de tercera generación [C3G] emergen como un desafío crítico en la interfaz salud humana-animal. La inocuidad de los animales de consumo junto con la RAM se han convertido en una preocupación en el enfoque de Una Salud. El objetivo de la investigación fue caracterizar aislamientos de Escherichia coli resistentes a C3G de origen porcino y humano para ver la asociación entre estas poblaciones.
Materiales y métodos
De una base de datos que comprende 1408 aislamientos de E. coli provenientes de materia fecal de humano (425) y porcino (983), se seleccionaron 88 cepas resistentes a ceftazidima [CAZ] y cefotaxima [CTX]. Los aislamienos de E. coli de porcinos (25/88) se obtuvieron de un muestreo realizado en una granja semitecnificada ubicada en Jiutepec, Morelos, México y los aislamientos de origen humano (63/88) provienen de tres centros de salud (CS) colindantes a la granja (CS Jiutepec, CS Huizachera, CS Calera Chica) y del Hospital General de Cuernavaca “José G. Parres” en Cuernavaca, Morelos, México. La resistencia a C3G se confirmó mediante concentración mínima inhibitoria (MIC) usando la técnica de microdilución en caldo (CLSI, 2023). La identificación de genes de resistencia a C3G (blaCTX-M, blaSHV, blaTEM) (Saleem et al., 2022) de factores de virulencia (eae, stx1, stx2, elt, est, ipaH, aat, daa) (Tamayo-Legorreta, 2020) y de filogrupos (A, B1, B2, C, D) (Clermont et al., 2000) se realizón por PCR punto final. Por electroforesis en campos pulsados (PFGE) se determinó la clonalidad de las cepas.
Resultados
Se analizaron 88 cepas de E. coli multidrogo resistentes [MDR]. De estas, 63 cepas de origen humano (71,6%) mostraron al menos un marcador de patogenicidad, siendo los tipos más comunes DAEC (9,1%) y EIEC (4.55%). En cerdos, no se aislo ningún patotipo de E. coli. Los genes de virulencia más comunes en humanos fueron ipaH (4,55%) y daa (9,1%). En cerdos, todas las cepas fueron comensales, es decir, no tenían genes de virulencia. La resistencia a los antibióticos CAZ y CTX fue alta en ambos grupos: >70% en humanos y >25% en cerdos. El 78,4% (69/88) fueron identificadas como productoras de BLEE predominando el gen de resistencia blaCTX-M. Las cepas de E. coli se agruparon en 18 perfiles de resistencia antimicrobiana, 86 cepas (97,73%) fueron MDR, de las cuales 68 (77,27%) fueron resistentes a seis, siete y ocho familias de antibióticos. Por otra parte, las cepas de E. coli son geneticamente diferentes, lo que significa que hay una mayor diversidad de tipos de E. coli en los humanos comparado con los cerdos. En los aislamientos de humanos se observaron los filogrupos A (23,86%), B1 (7,95%), B2 (5,68%) y D (34,1%); y en los porcinos encontramos los grupos A (22,73%), B1 (1,14%) y D (4,55%). Los filogrupos A y B1 corresponden a cepas de E. coli comensales mientras que B2 y D son un indicador de contaminacion fecal humana y se asocian a E. coli patógena. El análisis de PFGE comprobó que existe una diversidad genética entre las cepas de cerdos y humanos mostrando que no están clonalmente relacionadas.
Todos los resultados se resumen en la Figura 1. Los Dendogramas de la Electroforésis en Campos Pulsados (PFGE) de cepas de E.coli aisladas de materia fecal de cerdos (A) y humanos (B) muestran la diversidad y los patrones de resistencia (Figura 1). Las características fenotípicas y genotípicas detallan la producción de BLEE (CTX-M) y su asociación con la multiresistencia antimicrobiana en clonas de E. coli en ambas especies, información crucial para decisiones clínicas y vigilancia epidemiológica.
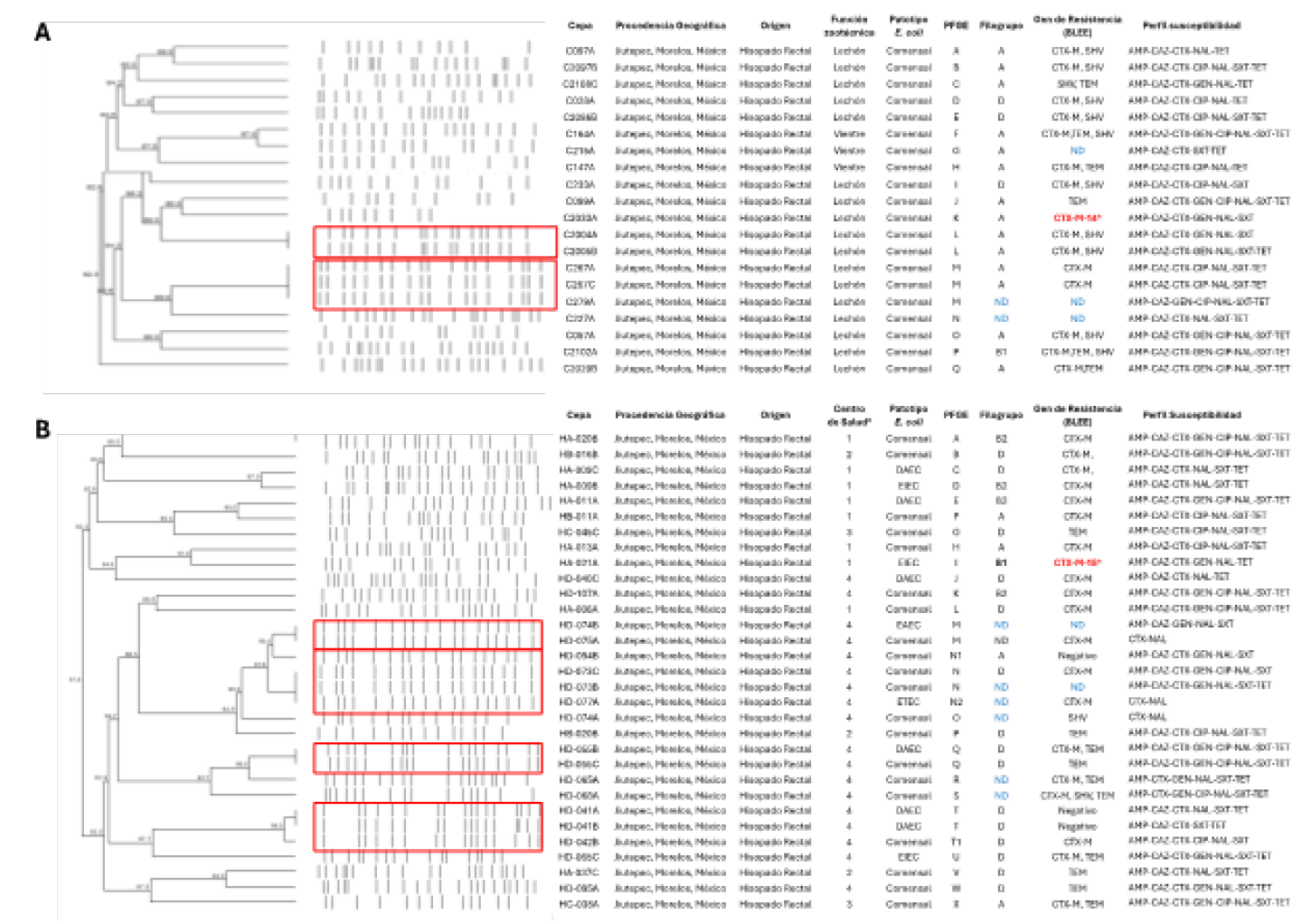
Figura 1. Relación clonal por PFGE de 31 aislamientos representativos de E. coli aislada de heces de humano y 20 aislamientos representativos aislados de heces de cerdo. Los antibióticos analizados son: AMP= Ampicilina, CAZ= Ceftazidima, CTX= Cefotaxima, GEN= Gentamicina, CIP= Ciprofloxacino, NAL= Ácido nalidíxico, SXT= Sulfametoxazol/trimetoprim, TET= Tetraciclina. ND= No determinado. a 1-CS Jiutepec, 2-CS Huizachera, 3- CS Calera Chica, 4-Hospital General de Cuernavaca “Dr José G. Parres, Cuernavaca, Morelos, México.*
* Los paneles en rojo muestran la agrupación de 11cepas representativas de humanos en 4 clústeres y 5 cepas representativas de cerdos en dos clústeres. El porcentaje de similitud entre las cepas se representó en un dendograma de homología utilizando el algoritmo UPGMA.
Conclusiones
Los resultados obtenidos subrayan la importancia de monitorear y controlar la RAM y la patogenicidad de E. coli para prevenir y tratar infecciones adecuadamente. La presencia de BLEE en animales de consumo humano, como el cerdo, potencialmente puede seleccionar bacterias resistentes que posteriormente logren transferirse de forma horizontal al consumidor (humano) a través del contacto directo y por la cadena alimenticia. Es fundamental abordar este problema de manera integral mediante prácticas de saneamiento, uso responsable de antibióticos en la medicina humana y veterinaria, y campañas de salud pública sobre los riesgos de la RAM, para minimizar la resistencia en la interfaz de Una Salud.
Downloads
Metrics
Article Details

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
The authors who publish in Siembra know and accept the following conditions:
- Authors retain the copyright and grant Siembra the right of first publication of the work, under the Creative Commons Attribution License. Third parties are allowed to use what has been published as long as they refer to the author or authors of the work and its publication in this journal.
![]() This content is licensed under a Creative Commons Attribution-Noncommercial 4.0 International (CC BY-NC 4.0).
This content is licensed under a Creative Commons Attribution-Noncommercial 4.0 International (CC BY-NC 4.0).
- Authors maintain the copyright and guarantee Siembra the right to publish the manuscript through the channels it considers appropriate.
- Authors may establish on their own additional agreements for the non-exclusive distribution of the version of the work published in Siembra, acknowledging their initial publication in the same, such as in institutional repositories.
- Authors are authorized to disseminate their work electronically once the manuscript is accepted for publication.
References
Clermont, O., Bonacorsi, S., y Bingen, E. (2000). Rapid and simple determination of the Escherichia coli phylogenetic group. Applied and Environmental Microbiology, 66(10), 4555-4558. https://doi.org/10.1128/AEM.66.10.4555-4558.2000
Clinical and Laboratory Standards Institute [CLSI]. (2023). Performance Standards for Antimicrobial Susceptibility Testing (33rd ed.). CLSI supplement M100. CLSI.
Saleem, M., Rashid, F., Liaqat, I., Liaqat, I., Ulfat, M., Sultan, A., Faiz, M., Eijaz, S., y Bibi, A. (2022). Phenotypic and Molecular Characterization of CTX-M Type B-Lactamases in gram negative bacterial strains isolated from hospitals, Lahore, Pakistan. Journal of Oleo Science, 71(6), ess22041. https://doi.org/10.5650/jos.ess22041
Tamayo-Legorreta, E. M., García-Radilla, A., Moreno-Vázquez, E., Téllez-Figueroa, F., y Alpuche-Aranda, C. M. (2020). Diarrheagenic Escherichia coli pathotypes isolated from a swine farm in a region of Morelos state, Mexico. Salud Pública de México, 63(1), 34-41. https://doi.org/10.21149/11268

