Filogenia y Resistencia a Antibióticos en Micobacterias No Tuberculosas: Un Enfoque Integrado para el Manejo Clínico
Main Article Content
Abstract
Introducción
Las micobacterias no tuberculosas [MNT] representan un grupo diverso de patógenos que requieren un manejo clínico específico debido a su variabilidad en resistencia a antibióticos. Este estudio se centra en la identificación filogenética de especies de MNT mediante el análisis de los genes 16S rRNA, hsp65 y rpoB, y su correlación con perfiles de resistencia a antibióticos.
Materiales y métodos
Se recolectaron 57 aislados clínicos de MNT entre 2019 y 2022. Se amplificaron y secuenciaron los genes 16S rRNA, hsp65 y rpoB. Las secuencias obtenidas se alinearon y se analizaron filogenéticamente utilizando el software PAUP 4.0. Además, se realizaron pruebas de susceptibilidad a antibióticos mediante microdilución en caldo (Sensititre RapidMyco y SlowMyco para MNT crecimiento rápido y lento, respectivamente).
Resultados
El análisis filogenético reveló la presencia de varios clados, destacando M. abscessus, M. avium, M. fortuitum, y M. intracellulare (Figura 1). M. abscessus mostró las tasas más altas de resistencia, con un 80% de resistencia a doxiciclina y un 50% a moxifloxacino. En contraste, M. avium presentó un perfil de resistencia más bajo. M. fortuitum e M. intracellulare mostraron un perfil intermedio, con tasas significativas de resistencia a trimetoprima/sulfametoxazol y ciprofloxacino. La variabilidad en la resistencia se correlacionó con las diferencias en la estructura genética de los clados. Los resultados indicaron que el gen rpoB mostró una mayor variabilidad, lo que permitió una identificación más precisa a nivel de especies e incluso subespecies. Además, se observó una correlación entre el perfil de resistencia y el contenido de G-C%, lo que sugiere que las variaciones en el gen rpoB pueden estar asociadas con la resistencia a los antibióticos.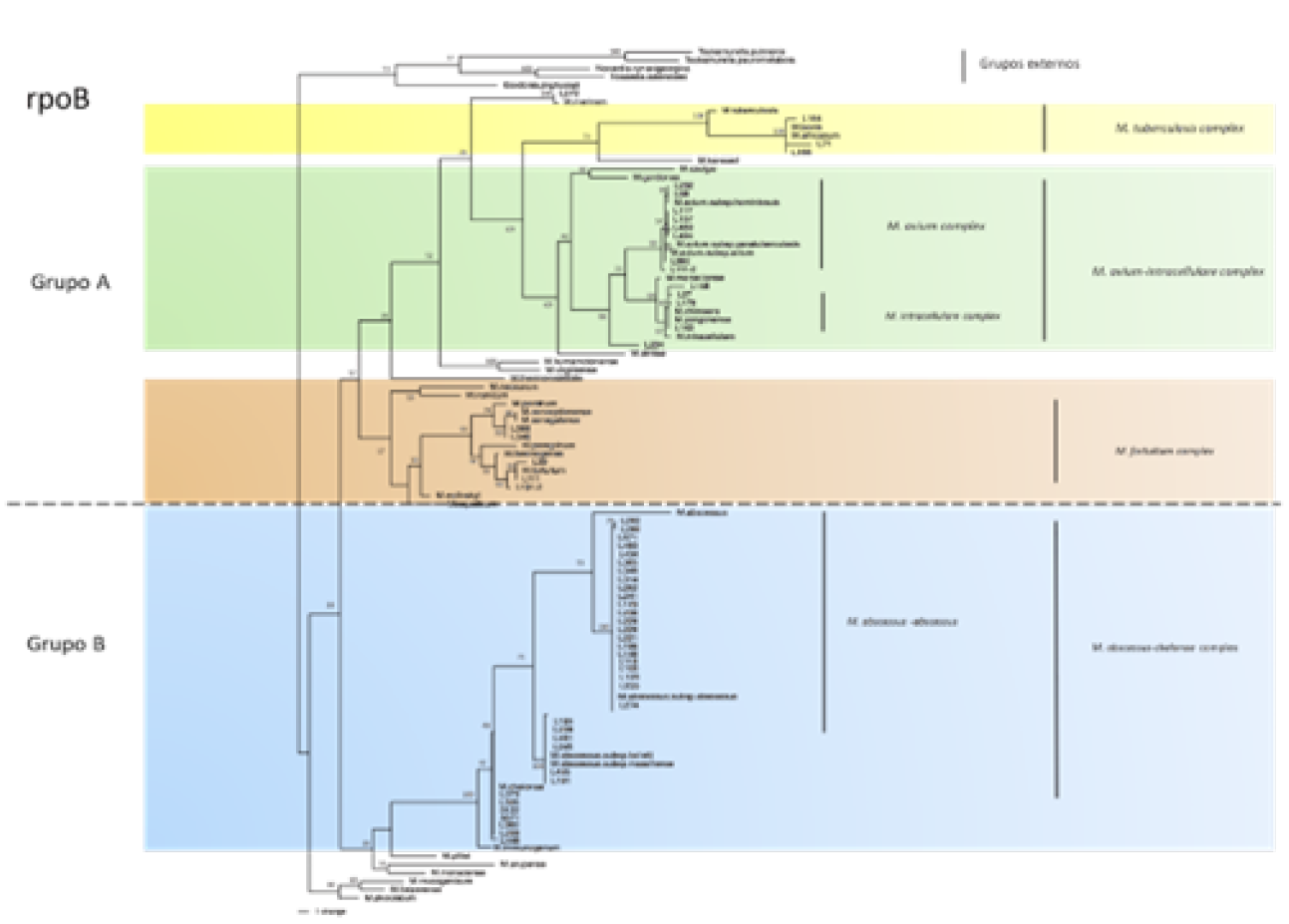
Figura 1. Análisis filogénetico del gen rpoB de especies de Mycobacterium
En micobacterias de crecimiento rápido [MCR], la amikacina fue el fármaco más efectivo, con una tasa de resistencia del 0% seguido por claritromicina con una resistencia del 7.5%. Se observaron altas tasas de resistencia a moxifloxacino (50%), linezolid (42,5%), doxiciclina (80%), imipenem (62,5%), tobramicina (67,5%), y trimetoprima/sulfametoxazol (62,5%). En micobacterias de crecimiento lento [MCL], no se observó resistencia a claritromicina, pero se registraron tasas de resistencia a isoniazida (50%), estreptomicina (50%), rifampicina (14,3%), etambutol (14,3%), y etionamida (28,6%). La resistencia a amikacina fue del 14,3%.
Conclusiones
La correlación entre la filogenia y la resistencia sugiere que la evolución de la resistencia puede estar vinculada a cambios genéticos específicos en los marcadores analizados. Las diferencias en la resistencia observadas entre los clados resaltan la importancia de enfoques personalizados en el tratamiento de infecciones por micobacterias.
Downloads
Metrics
Article Details

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
The authors who publish in Siembra know and accept the following conditions:
- Authors retain the copyright and grant Siembra the right of first publication of the work, under the Creative Commons Attribution License. Third parties are allowed to use what has been published as long as they refer to the author or authors of the work and its publication in this journal.
![]() This content is licensed under a Creative Commons Attribution-Noncommercial 4.0 International (CC BY-NC 4.0).
This content is licensed under a Creative Commons Attribution-Noncommercial 4.0 International (CC BY-NC 4.0).
- Authors maintain the copyright and guarantee Siembra the right to publish the manuscript through the channels it considers appropriate.
- Authors may establish on their own additional agreements for the non-exclusive distribution of the version of the work published in Siembra, acknowledging their initial publication in the same, such as in institutional repositories.
- Authors are authorized to disseminate their work electronically once the manuscript is accepted for publication.

